Hauptinhalt
Topinformationen
Forschungsfeld B
Visualisierung und Manipulation von Mikrokompartimenten mit höchster Auflösung
Koordinator: Jacob Piehler
Im Rahmen von Forschungsfeld B sollen höchstauflösende Mikroskopietechniken weiterentwickelt werden. Ihre Anpassung an Untersuchungsverfahren dynamischer Mikrokompartimente und die wirkungsvolle Kombination komplementärer Bildgebungstechniken sind zentrale Anliegen dieses Forschungsfeldes. Dies erfordert
- die Bereitstellung modernster hochauflösender Fluoreszenzmikroskopietechniken
- Einzelmoleküllokalisationsbasierte Fluoreszenztechniken zur höchstauflösenden, dreidimensionalen Bildgebung, die dann zur Untersuchung von Mikrokompartimenten in verschiedenen Zelltypen adaptiert werden
- Multiphotonenmikroskopische Techniken insbesondere mit Bezug auf die Bildgebung auf der Basis verschiedener Nichtlinearer Optik (NLO)-Nanomaterialien
- elektronenmikroskopische Techniken zur dreidimensionalen ultrastrukturellen Bildgebung und zur Lokalisation von Proteinkomplexen sowie für korrelative Mikroskopie
Beteiligte Wissenschaftlerinnen und Wissenschaftler
- Brandt, Biologie
- Haase, Chemie
- Heinisch, Biologie
- Hensel, Biologie
- Holthuis, Biologie
- Imlau, Physik
- Kerkhoff, Humanwissenschaften
- Kost, Biologie
- Kurre, Biologie
- Paululat, Biologie
- Piehler, Biologie
- Rosemeyer, Chemie
- Schmitz, Biologie
- Touma, Biologie
- Ungermann, Biologie
- You, Biologie
- Zachgo, Biologie
Hoch und höchstauflösende Mikroskopietechniken im CellNanOs
| Technik | Verantwortlichkeit |
|---|---|
| Konfokale Mikroskopie inkl. MPE, FLIM, FCS | Brandt, Hensel, Piehler |
| Spinning disk Mikroskopie | Hensel |
| Dekonvolutionsmikroskopie | Ungermann |
| Einzelmolekül-Lokalisationsmikroskopie (PALM/STORM/TALM) | Piehler |
| Rasterkraftmikroskopie mit Fluoreszenzmikroskopie | Hensel, Piehler |
Multiphotonen-Mikroskopietechniken | Imlau |
| Upconversion-Mikroskopie | Haase, Piehler |
| Transmissionselektronenmikroskopie | Paululat, Hensel |
| Rasterelektronenmikroskopie | Paululat |
Mechanismen der Entwicklung und Degeneration von Nervenzellen
Thema der AG Brandt ist die Aufklärung der molekularen Mechanismen der Entwicklung, des Alterns und der Degeneration von Nervenzellen. Einer der Schwerpunkte der Arbeitsgruppe besteht in der Entwicklung und dem Einsatz quantitativer „live cell imaging“ Methoden, um die Dynamik von Komponenten des neuronalen Zytoskeletts zu verfolgen. Die Analyse erfolgt in zellulären Modellen und organotypischen Hirnkulturen. Darüber hinaus entwickelt die Arbeitsgruppe neue Mausmodelle, um Mithilfe von konfokaler und 2-Photonen-Mikroskopie morphologische Veränderungen von Nervenzellen in einem systemischen Kontext zu bestimmen.
Direkt zur Arbeitsgruppe Brandt, Biologie
Synthese anorganischer Nanokristalle
Die AG Haase beschäftigt sich mit der chemischen Synthese von funktionalen anorganischen Nanokristallen und deren struktureller und optischer Charakterisierung. In jüngerer Zeit wurden vor allem Upconversion-Nanopartikel untersucht. Hierzu werden neue Synthesestrategien entwickelt, um Partikel mit enger Partikelgrößenverteilung und definiert einstellbaren Partikelgrößen herstellen zu können. Einen weiteren Schwerpunkt stellen Partikel mit unterschiedlichen Kern-Schale-Strukturen dar, in denen sich die für die optischen Eigenschaften maßgeblichen Dotierungsatome in genau definierten Bereichen innerhalb der Partikel befinden.
Direkt zur Arbeitsgruppe Haase, Chemie
Signaltransduktion im Hefe-Zellintegritätsweg
Die AG Heinisch untersucht die Signaltransduktion mechanischer Sensorproteine in Verbindung mit Zellwandsynthese und Zytokinese bei Hefen (Saccharomyces cerevisiae und Kluyveromyces lactis). Die Gruppe verwendet dabei sowohl klassisch-genetische Ansätze (Mutantenscreens, Tetradenanalyse) als auch moderne molekularbiologische Techniken (Klonierungen über in vivo-Rekombination, genomische Tags etc.). In Zusammenarbeit mit anderen Arbeitsgruppen werden sowohl Verfahren für den Nachweis einzelner Sensoren mit Hilfe der AFM entwickelt, als auch Methoden die Dynamik der Sensoren in den Mikrokompartimenten der Plasmamembran zu untersuchen.http://www.biologie.uni-osnabrueck.de/arbeitsgruppen/genetik.html
Direkt zur Arbeitsgruppe Heinisch, Biologie
Interaktion pathogener Bakterien mit Wirtszellen

Dreidimensionale, höchstaufgelöste Visualisierung des Typ III Sekre-tionssystems in Salmonella mittels dSTORM (rechts) (AG Hensel, unveröffentlicht).
Im Mittelpunkt der Forschung der AG Hensel stehen die zellulären und molekularen Interaktionen bei bakteriellen Infektionen.
Am Modellsystem des Erregers Salmonella enterica werden molekulare Mechanismen der Umsteuerung eukaryontischer Wirtszellen bei der Adhäsion, Invasion und intrazellulären Lebensweise analysiert. Hierbei kontrollieren bakterielle Virulenzfaktoren das Aktin-Zytoskelett der Wirtszelle, wie auch den endosomalen Transport. Die Gruppe untersucht mittels Lebendzell- und Elektronenmikroskopie, sowie korrelativer und höchstauflösender Bildgebung die spatiotemporale Dynamik diese Vorgänge.
Direkt zur Arbeitsgruppe Hensel, Biologie
Dynamik und Homöostase der Membranlipide
Die AG Holthuis untersucht die Grundprinzipien von Transport und Homöostase der Membranlipide. Das Forschungsgebiet umfasst die zugrundeliegende Proteinmaschinerie aus Lipid-Konvertern, -Sensoren und -Flippasen mitsamt möglicher Fehlfunktionen, die systemische Ausfälle und letztlich Krankheiten bedingen. Im diesem Zusammenhang entwickelt die AG Methoden zum Visualisierung der lokalen Lipid- Pools im lebenden Zellen (Semisynthetische Lipid-Sensoren), Manipulation der Zellulare Lipid-Flusses (Umschaltbare Enzymen/Transporter), und Identifizierung von funktionellen Protein-Lipid Wechselwirkungen in Zellmembranen (Bifunktionale Lipid-Technologie).
Direkt zur Arbeitsgruppe Holthuis, Biologie
Ultrakurzzeitphysik - Visualisierung und Manipulation mit Femtosekunden-Lasermikroskopie

Erzeugung von ultraviolettem Licht durch Beleuchtung mit infraroten Femtosekunden Laserpulsen in harmonischen Niobat-Nanokristallen (unveröffentl. Erg.)
Die Forschungsgruppe Imlau untersucht neue Methoden zur Steuerung und Kontrolle nanophotonischer Sonden in zellulären Mikrokompartimenten. Ein besonderer Fokus liegt auf der lokalen Erzeugung von ultravioletter Strahlung (< 400 nm) mittels harmonischer Nanopartikel zur hochaufgelösten (< 100 nm) Photomanipulation in Zellen. Hierzu studieren wir Effizienzen und neuartige physikalische Prozesse bei der UV-Lichterzeugung in maßgeschneiderten harmonischen Nanopartikeln unter Verwendung von Femtosekunden-Laserpulsen, z.B. durch nichtlinear-optische Resonanz.
Dafür nutzen wir unsere umfassenden Möglichkeiten, Femtosekunden-Laserpulse in Hinblick auf Formung und komplexer zeitlicher Abfolge sowie Wellenlänge zu kontrollieren.
Direkt zur Arbeitsgruppe Mirco Imlau, Physik
Molekulare Mechanismen der Immunaktivierung - S100A8/A9 Proteine: Rezeptoren und Signaltransduktion
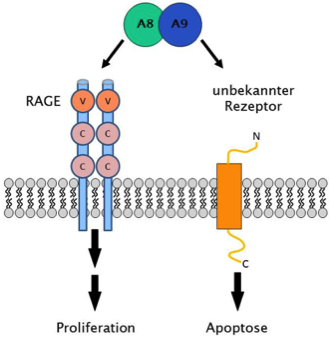
Über verschiedene, teilweise noch unbekannte Rezeptoren kann S100A8/A9 in Zielzellen sowohl die Proliferation als auch die Apoptose induzieren (Ghavami et al., BBA 2008)
Die AG Kerkhoff untersucht die Signaltransduktion des Calcium-bindenden Proteinkomplexes S100A8/A9 nach Bindung an unterschiedliche Rezeptoren, die sowohl die Proliferation als auch die Apoptose in den Zielzellen (Tumorzellen) induzieren können.
Die Gruppe verwendet dabei moderne molekularbiologische und biochemische Techniken, um Protein-Protein-Wechselwirkungen zu identifizieren, zu analysieren und ihre raum/zeitliche Dynamik durch höchstauflösende Bildgebung aufzuklären.
Im Einzelnen sollen die Rezeptoren charakterisiert, die Protein-Protein-Interaktionen auf der Plasmamembran quantifiziert und die Signaltransduktion analysiert werden.
Direkt zur Arbeitsgruppe Kerkhoff, Humanwissenschaften
Metabolische Stoffaustauschinteraktionen zwischen Bakterien - Kontaktabhängige Wechselwirkungen zwischen Bakterien
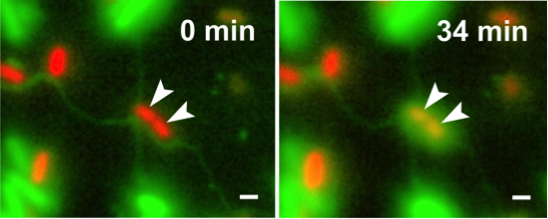
Escherichia coli (grüne Zellen) nutzt interzelluläre Nanokanäle um zytoplasmatische Bestandteile auf Acinetobacter baylyi (rote Zellen) zu übertragen (Pande et al., Nat Commun 2015)
Die Forschungsgruppe Kost untersucht kontaktabhängige metabolische Interaktionen zwischen verschiedenen Bakterienzellen. Ein Schwerpunkt liegt auf der Erforschung lipidbasierter Nanokanäle, die von auxotrophen Bakterien gebildet werden, um Aminosäuren aus dem Zytoplasma anderer Bakterienzellen zu erhalten. Ziel ist es, die molekularen und physiologischen Grundlagen interzellulärer Nanokanäle zu identifizieren, sowie zu einem besseren Verständnis ihrer Funktion beizutragen.
Dafür werden Nanokanal-bildende Zellen mit Hilfe von hoch- und höchstauflösenden Licht- und Elektronenmikroskopen sichtbar gemacht, um die dem Metabolittransport zugrundeliegende molekulare Maschinerie besser zu verstehen.
Direkt zur Arbeitsgruppe Kost, Biologie
Höchstauflösende Fluoreszenzmikroskopie - Multimodale Fluoreszenzmikroskopie mit höchster räumlicher und zeitlicher Auflösung
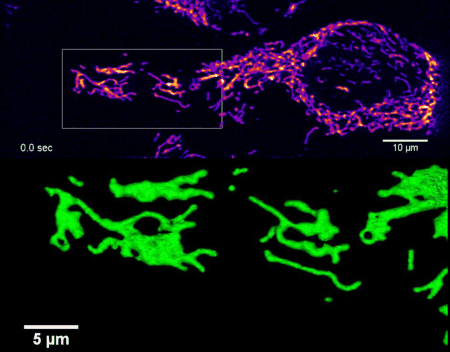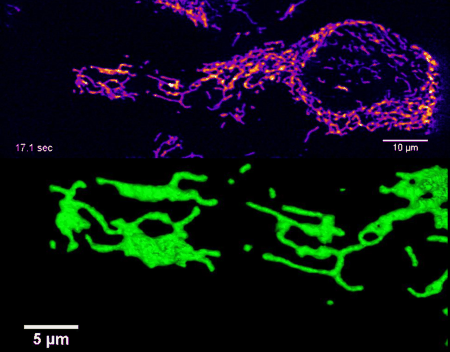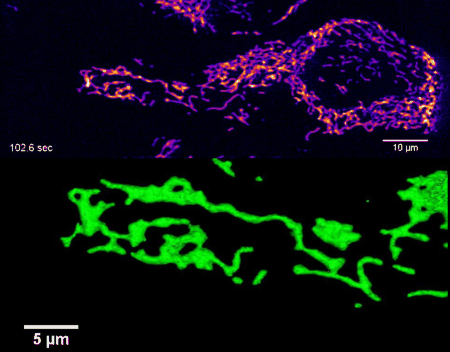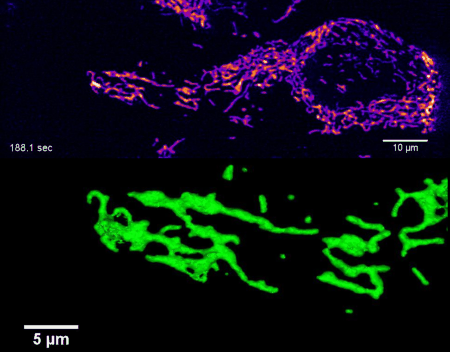
Hochaufgelöste vierdimensionale Bildgebung des mitochondrialen Netzwerks in lebenden HeLa-Zellen mittels Lattice Light Sheet Mikroskopie (nicht-publizierte Ergebnisse).
Die Arbeitsguppe Rosemeyer konzipiert und etabliert gemeinsam mit Arbeitsgruppen aus der Biologie, Chemie und Physik neuartige Fluoreszenzmikroskopietechniken für die Lebendzellmikroskopie. In enger Zusammenarbeit mit den Arbeitsgruppen aus der Biologie werden Arbeitsabläufe und Protokolle für verschiedenste biologische Proben entwickelt.
Ziel ist die Bereitstellung von maßgeschneiderten Mikroskopiesystemen, die einem breiten Nutzerkreis die Anwendung neuester multimodaler Bildgebungsmethoden mit höchster zeitlicher und räumlicher Auflösung ermöglichen. Der Fokus liegt auf der Mehrfarben-Einzelmolekül-Lokalisationsmikroskopie sowie der Kombination von strukturierter Beleuchtung und der Lichtblattmikroskopie (lattice light-sheet mikroscopy).
Direkt zur Arbeitsgruppe Kurre, Biologie
Extrazelluläre Matrix und Organogenese
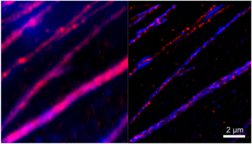
Co-Organisation von Pericardin- (rot) und Collagenfilamenten (blau) in der extrazellulären Matrix im Herzgewebe von Drosophila mittels dSTORM (rechts) im Vergleich zu beugungsbegrenzte Bildgebung (AG Paululat, AG Piehler, unveröffentl.)
Die AG Paululat befasst sich mit den molekularen Mechanismen der Herz- und Muskeldifferenzierung im Modellorganismus Drosophila. Insbesondere die Assemblierung und Funktion der extrazellulären Matrix des Herzens wird mit Hilfe genetischer, molekularbiologischer und mikroskopischer Methoden analysiert.
Neben der funktionellen Charakterisierung von Proteinen, die am Matrixaufbau beteiligt sind, hier speziell Collagen-ähnliche Proteine und Metalloproteasen, liegt ein Forschungsschwerpunkt der Gruppe auf der Nutzung und Optimierung bildgebender Verfahren die der Analyse der Matrixassemblierung und ihres Aufbaus dienen.
Direkt zur Arbeitsgruppe Paululat, Biologie
Signalvermittlung über biologische Membranen - Raum/zeitliche Dynamik von Signalkomplexen
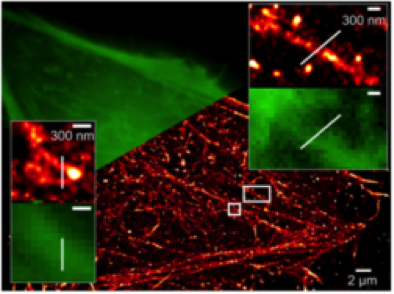
Höchstaufgelöste Abbildung des kortikalen Aktinzytoskeletts in lebenden Zellen mittels dSTORM (Wilmes et al., Angew. Chem., 2012)
Die AG Piehler untersucht die Assemblierung und Dynamik von Signalkomplexen in der Plasmamembran lebender Zellen mittels Einzelmolekülmikroskopie.
Zielproteine werden über posttranslationale Konjugationsmethoden mit photostabilen Farbstoffen oder lumineszenten Nanopartikeln markiert (siehe Forschungsfeld A) und ihre Koorganisation bzw. Dimerisierung über Einzelmoleküllokalisation analysiert. Ziel ist es, die Rolle der submikroskopischen Kompartimentierung der Plasmamembran für die raum/zeitliche Dynamik von Zytokinrezeptorkomplexen aufzuklären und mit der Signalspezifizität zu korrelieren. Mittels Lichtblattmikroskopie soll künftig die Signalweiterleitung auch an endosomalen Vesikeln in der Zelle verfolgt werden.
Direkt zur Arbeitsgruppe Piehler, Biologie
Bioorganische Synthese von Nucleolipiden als "Antitumor Small Molecules"
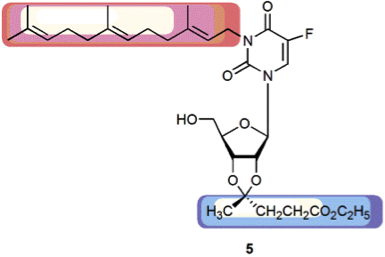
Neuartiges Nukleolipid mit signifikanter cytotoxischer Aktivität gegenüber verschiedenen humanen Tumorzelllinien, jedoch keiner Aktivität gegenüber PMA-differenzierten THP-1 Makrophagen (Knies et al., Chem Biodivers 2015)
Die AG Rosemeyer bearbeitet die bioorganische Synthese von Nucleolipid-Hybridmolekülen aus kanonischen Nucleosiden sowie aus Nucleosid-Antimetaboliten und natürlichen und synthetischen Lipiden. Diese werden im Hinblick auf ihre cytostatische/ cytotoxische Aktivität gegenüber a) humanen Tumorzelllinien des NCI 60 Panels sowie b) humanen Glioblastom Tumorzellen untersucht.
Durch Vergleich ihrer Anti-Tumor-Wirkungen und den konformationellen Parametern der neuartigen Verbindungen mittels detaillierter Mehr-Kern-NMR-Spektroskopie sollen quantitative Struktur-Aktivitäts-Beziehungen (QSAR-Studie) gewonnen werden.
Direkt zur Arbeitsgruppe Rosemeyer, Chemie
Dynamik und Assemblierung von Zytoskelett-organisierenden Komplexen
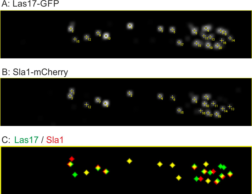
Co-lokalisationsanalyse mittels Detektion und zeitlicher Verfolgung von Endozytoseereignissen in Ashbya gossypii am Beispiel der Regulatoren Las17 und Sla1.
Die AG Schmitz beschäftigt sich mit Veränderungen des Zytoskeletts während des polaren Wachstums und der Morphogenese. Dabei werden Pilzzellen als Modellsystem für schnelles und gerichtetes Wachstum verwendet. Untersucht werden dabei sowohl Vorgänge der frühen Endozytose als auch der Wandel des Spindelpolkörpers von einem Mikrotubuli- zu einem Aktinorganisationszentrum während der Sporulation.
Bei beiden Projekten kommen in erster Linie lichtmikroskopische Methoden zur Beobachtung und Quantifizierung von dynamischen Prozessen in lebenden Zellen zum Einsatz.
Die präzise Auswertung dieser dynamischen Prozesse wird erst durch eine in Zusammenarbeit mit dem CALMOS entwickelte Software ermöglicht. Zusätzlich werden in diesem Projekt auch optogenetische Methoden entwickelt und getestet.
Direkt zur Arbeitsgruppe Schmitz, Biologie
Molekulare Grundlagen des Verhaltens - Wechselwirkung von Stress und Energiemetabolismus in klinisch relevanten Tiermodellen
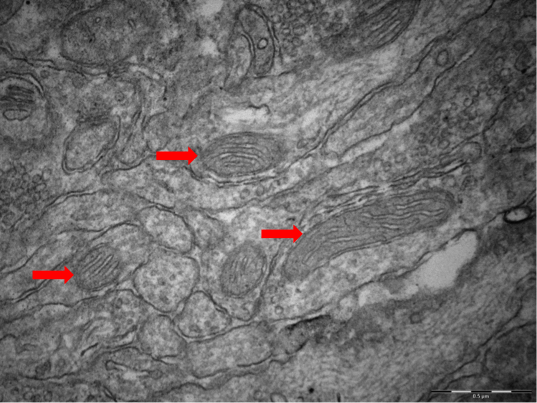
Elektronenmikroskopische Aufnahme von Mitochondrien im Hippocampus einer "High Reactivity-Maus"
Die Forschungsansätze der AG Touma gehen der Frage nach, wie Stress und Änderungen im neuronalen und peripheren Energiemetabolismus zusammenwirken und welche Rolle sie bei der Ätiologie von Krankheiten spielen. Studien belegen bei Patienten und in Tiermodellen psychiatrischer Erkrankungen mechanistische Zusammenhänge von Störungen der Stresshormonregulation und zellulären Veränderungen, die letztlich zu einer beschleunigten Zellalterung beitragen.
Die AG verwendet ein Stress-Reaktivitäts-Mausmodell, um die zugrundeliegenden Risikofaktoren und Pathomechanismen aufzuklären. Dafür nutzen wir die Visualisierung mit höchster Auflösung und anschließender Datenprozessierung mit modernsten Methoden, um in unseren Tiermodellen zellbiologische Determinanten der Stressregulation aufzuspüren.
Direkt zur Arbeitsgruppe Touma, Biologie
Membrandynamik im endosomalen Weg
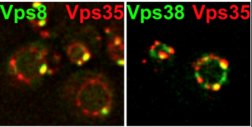
Verteilung endosomaler Proteine relativ zueinander. Die entsprechenden Hefeproteine wurden mit GFP und mCherry Markern genomisch markiert. Die ringartige Struktur ist die Vakuole der Hefe (Arlt et al., Mol Biol Cell, 2015).
Die AG Ungermann ist an der Fusion von Membranen im endolysosomalen Weg der Hefe und höherer Eukaryonten interessiert. Die Gruppe hat Protokolle zur Analyse zur Visualisierung von Mikrokompartimenten auf Vakuolen, und zur Verfolgung und Manipulation des endozytischen Transports entwickelt.
Höchstauflösende Strukturen auf beiden Kompartimenten werden mit ultrastrukturellen Techniken untersucht. Die Gruppe entwickelt dabei neuartige Techniken zur schnellen Analyse der raumzeitlichen Veränderung von endosomalen und vakuolären Strukturen in Zusammenarbeit mit Gruppen des CellNanOs.
Direkt zur Arbeitsgruppe Ungermann, Biologie
Biomoleküle an Grenzflächen/Organisation und Dynamik des kanonischen Wnt Signalkomplexes
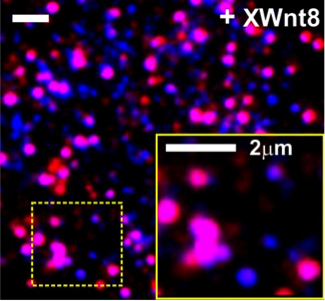
Einzelmolekül-Mikroskopie offenbart die Wnt-induzierte Assemblierung von Signalosomen in der Plasmamembran lebender HeLa-Zellen (unpublizierte Ergebnisse).
Die Nachwuchsgruppe You untersucht die raum/zeitliche Organisation des kanonischen Wnt-Signalwegs, einem zentralen Angelpunkt in der Regulation von Zelldifferenzierung und embryonaler Entwicklung. Das molekulare Wechselspiel der zahlreichen Proteine, die an der Bildung des sogenannten „Wnt-Signalosoms“ beteiligt sind, ist derzeit noch unklar.
Mittels Mehrfarben-Einzelmolekülmikroskopie sowie mikrostrukturierter Immobilisierung von Proteinen in der Plasmamembran lebender Zellen möchten wir die molekularen und zellulären Determinanten bestimmen, die die Assemblierung, Dynamik und Signalaktivierung von Wnt Signalosomen bestimmen.
Direkt zur Arbeitsgruppe You, Biologie
Pflanzliche Entwicklungsgenetik - Transkriptionsregulation im Zellkern
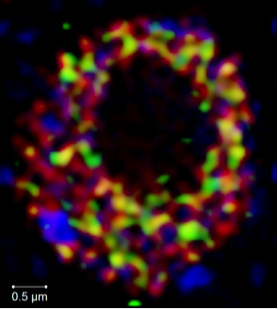
Auflösung der subnuklearen Lokalisation von ROXY1 (grün), der RNA Polymerase II (rot) und des Heterochromatins (blau) mittels strukturierter Beleuchtung (SIM) (unveröffentlichte Ergebnisse).
Die AG Zachgo untersucht die Funktion von ROXYs, Landpflanzen-spezifischen Glutaredoxinen (GRX), die für die Anlage und Differenzierung von Pflanzenorganen essentiell sind. ROXYs wechselwirken mit den bZIP TGA Transkriptionsfaktoren, die von ROXYs posttranslational im Zellkern modifiziert werden könnten.
Mittels hoch- und höchstauflösender Mikroskopie werden Protein-Protein-Wechselwirkungen in lebenden und fixierten Zellen untersucht. Die dreidimensionale Anordnung dieser Proteine im Zellkern soll mit Lichtblatt-SIM untersucht werden, um die Dynamik transkriptionaler Interaktionsnetzwerke in nuklearen Mikrokompartimenten aufzuklären.
Direkt zur Arbeitsgruppe Zachgo, Biologie

