Hauptinhalt
Topinformationen
Qualitative und quantitative Analytik von Zusammensetzung und Wechselwirkung
Koordinator: Heinz-Jürgen Steinhoff
Forschungsbereich C ist der Entwicklung und Bereitstellung von Methoden gewidmet, mittels derer die Protein- und Lipidzusammensetzung von Mikrokompartimenten analysiert sowie molekulare Wechselwirkungen identifiziert und quantifiziert werden können. Konkret umfaßt dies
- die Entwicklung und Adaptation massenspektrometrischer Protein- und Lipidanalytik in Kombination mit optischen und magnetischen Methoden zur selektiven Manipulation und Isolierung subzellulärer Proben
- den Ausbau quantitativer Wechselwirkungsanalytik mit gereinigten Proteinen und nativen zellulären Proben
- die Entwicklung von quantitativen Bindungstests zur Lokalisation und Quantifizierung miteinander wechselwirkender Bestandteile lebender Zellen
- die Anwendung und Weiterentwicklung strukturbiologischer Methoden bis hin zur Untersuchung von Proteinkonformationen und ihrer Dynamik in nativer Umgebung.
Beteiligte Wissenschaftlerinnen und -wissenschaftler
Qualitative und quantitative Interaktionsanalytik im CellNanOs
| Technik | Verantwortlichkeit |
|---|---|
| Massenspektrometrie (MALDI/ESI) | Ungermann |
| Quantitative Interaktionsanalytik an Oberflächen | Piehler |
| Rasterkraftmikroskopie | Piehler, Heinisch |
Proteinkristallisation | Kümmel |
| Elektronenspinresonanz-Spektroskopie | Steinhoff |
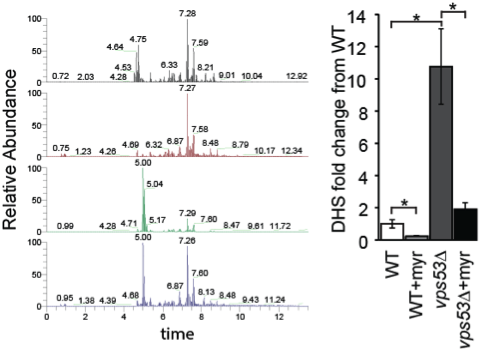
Massenspektrometrische Analyse der Sphingolipidzusammensetzung von Hefemutanten (Fröhlich et al., Elife 2015)
AG Florian Fröhlich, Biologie
Molekulare Membranbiologie - Mechanismen der Sphingolipid und Sterolhomöostase in Saccharomyces cerevisiae
Die Nachwuchsgruppe Fröhlich bearbeitet die Frage nach der Homöostase von Sphingolipiden und Sterolen im Modellorganismus Saccharomyces cerevisiae. Dazu wird die (Regulation der) Sortierung von Lipiden in intrazellulären Transportwegen untersucht. Neben den klassischen Methoden der Zellbiologie finden auch genetische Screens und die massenspektrometrische Analayse von Proteinen und Lipiden Verwendung. Ein detailliertes Verständnis dieser Prozesse bildet die Grundlage für die Untersuchung neurodegenerativer Erkrankungen, bei denen die Lipidhomöostase gestört ist.
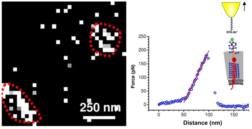
Kartierung von Sensor-Clustern an der Oberfläche von Hefezellen (links) und mechanische Eigenschaften von einzelnen Sensoren (rechts) mittels Rasterkraftmikroskopie und –spektroskopie (Dupres et al., Nat. Chem. Biol., 2009)
AG Jürgen Heinisch, Biologie
Signaltransduktion im Hefe-Zellintegritätsweg
Die AG Heinisch untersucht die Signaltransduktion mechanischer Sensorproteine in Verbindung mit Zellwandsynthese und Zytokinese bei Hefen (Saccharomyces cerevisiae und Kluyveromyces lactis). Die Gruppe verwendet dabei sowohl klassisch-genetische Ansätze (Mutantenscreens, Tetradenanalyse) als auch moderne molekularbiologische Techniken (Klonierungen über in vivo-Rekombination, genomische Tags etc.). In Zusammenarbeit mit anderen Arbeitsgruppen werden sowohl Verfahren für den Nachweis einzelner Sensoren mit Hilfe der AFM entwickelt, als auch Methoden die Dynamik der Sensoren in den Mikrokompartimenten der Plasmamembran zu untersuchen.
AG Michael Hensel, Biologie
Interaktion pathogener Bakterien mit Wirtszellen
Im Mittelpunkt der Forschung der AG Hensel stehen die zellulären und molekularen Interaktionen bei bakteriellen Infektionen. Am Modellsystem des Erregers Salmonella enterica werden molekulare Mechanismen der Umsteuerung eukaryontischer Wirtszellen bei der Adhäsion, Invasion und intrazellulären Lebensweise analysiert. Hierbei kontrollieren bakterielle Virulenzfaktoren das Aktin-Zytoskelett der Wirtszelle, wie auch den endosomalen Transport. Die Gruppe untersucht mittels Lebendzell- und Elektronenmikroskopie, sowie korrelativer und höchstauflösender Bildgebung die spatiotemporale Dynamik diese Vorgänge.
AG Christian Kost, Biologie
Metabolische Stoffaustauschinteraktionen
Die Arbeitsgruppe um Christian Kost erforscht kooperative Stoffaustauschinteraktionen zwischen verschiedenen Bakterienzellen. Hierzu müssen sowohl Metabolitkonzentrationen in- und außerhalb bakterieller Zellen, als auch die Aktivität der an ihrer Produktion beteiligten metabolischen Pfade erfasst werden. Die Forschungsgruppe erarbeitet Methoden zur präzisen Quantifizierung verschiedener Metabolite durch chemisch-analytische Verfahren oder genetisch-kodierte, intrazelluläre Biosensoren. Darüber hinaus werden sowohl die Flüsse durch die metabolischen Netzwerke von Bakterienzellen, als auch die Transkriptome einzelner Zellen bzw. ganzer Konsortien analysiert, um so metabolische Wechselwirkungen zwischen verschiedenen Organismen quantitativ zu verstehen.
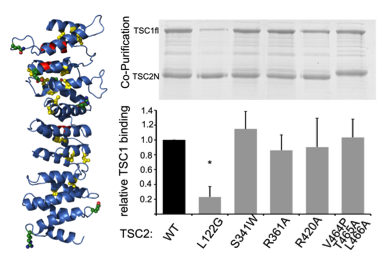
Durch die Strukturanalyse des Tumorsuppressors TSC2 (links) wurde der Mechanismus von pathogenen Mutationen, die zu Tuberöser Sklerose führen, aufgeklärt (rechts) (Zech et al., JBC 2016)
AG Daniel Kümmel, Biologie
Strukturbiologie molekularer Maschinen - Struktur und Dynamik von Proteinkomplexen in Signal- und Transportprozessen
Wie werden Transportprozesse und Signaltransduktion koordiniert und reguliert? Dazu untersucht die Emmy-Noether-Nachwuchsgruppe von Daniel Kümmel die Strukturen von Proteinen, die an diesen Prozessen beteiligt sind. Methodisch werden vor allem Röntgenkristallographie sowie ergänzende elektronenmikroskopische, biophysikalische und biochemische Ansätze genutzt. So kann die Proteinstruktur auf atomarer Ebene visualisiert und ihre Funktionsweise erklärt werden. Mit Hilfe eines derart detaillierten Verständnisses kann dann ihre Rolle im zellulären Kontext untersucht und gezielt manipuliert werden.

HRTEM Aufnahmen von {Mn4}-Komplexen an CNTs (links) bzw. C60 Molekülen in CNTs (Mitte). Rechts: AFM Aufnahme eines CNT-FETs mit skizzierter TERS Spitze
AG Carola Meyer, Physik
Elektronischer Transport - Ultra-sensitive Sensoren für molekulare und nanoskalige Phänomene
Die AG Meyer untersucht den elektronischen Transport in Kohlenstoffnanoröhren (CNTs), die mit Molekülen funktionalisiert sind. Im Fokus stehen molekulare Wechselwirkungen mit dem Ziel, ein tieferes Verständnis von Spinwechselwirkungen sowie molekularen Dynamiken in magnetischen und Biomolekülen zu entwickeln. Dazu gehört die Herstellung der Nano-Bauelemente inklusive Synthese und chemischer Funktionalisierung der Nanoröhren, sowie deren Charakterisierung mit modernsten mikroskopischen und spektroskopischen Verfahren. Der elektronische Transport wird mittels Quantentransport-, Rausch- und Magnetowiderstandsmessungen untersucht.
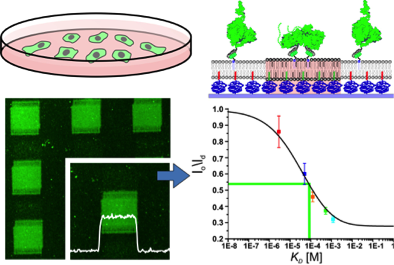
Quantifizierung niedrigaffiner Protein-Interaktionen mittels Anbindung an phasenseparierten, Polymer-unterstützten Membranen (Beutel et al., ACS Nano, 2015)
AG Jacob Piehler, Biologie
Signalvermittlung über biologische Membranen - Funktionale Organisation von Proteinen an Oberflächen
Die AG Piehler entwickelt Oberflächenmodifikationen zur funktionalen Anbindung von Proteinen. Eine wichtige Expertise bildet die Rekonstitution von Membranproteinen in Polymer-unterstützten Membranen. Durch Kombination von mikrostrukturierter Oberflächenfunktionalisierung mit optisch oder chemisch schaltbarer molekularer Erkennung entwickeln wir Methoden, um Proteine an Oberflächen und in Membranen räumlich und zeitlich kontrolliert zu organisieren. Kürzlich wurden diese Methoden erfolgreich auf Proteine in der Plasmamembran lebender Zellen ausgeweitet. Diese Oberflächenarchitekturen bieten einzigartige Möglichkeiten für die Anwendung von spektroskopischen und mikroskopischen Detektionsmethoden zur quantitativen Analytik biomolekularer Wechselwirkungen.

Rasterelektronenmikroskopisches Bild eines Fibroblasten auf einem Feld parallel angeordneter Polymernanostäbe (Grimm et al., J. Mater. Chem., 2010).
AG Martin Steinhart, Chemie
Mesoskopische Strukturbildung - Funktionale mikro- und nanostrukturierte Oberflächen
Die AG Steinhart untersucht das Design von in geeigneter Weise topographisch strukturierten funktionalen Oberflächen als synthetisch-biologische Schnittstellen. Die topographische Strukturierung kann mit einer nanoskopischen Feinstruktur als zusätzlicher funktionaler Hierarchieebene verbunden werden. Beispiele für Oberflächen, deren Topographie gezielt zur Kontrolle zellulärer Wechselwirkungen oder von Adhäsionseigenschaften eingestellt wird, sind Nanodraht- und Mikrokugelfelder. Beispiele für durch nanoskopische Größenskalen charakterisierte funktionale Hierarchieebenen/Feinstrukturen sind durchgehende kontinuierliche Porensysteme für den Transport von Flüssigkeit an die synthetisch-biologische Grenzfläche und Goldnanostrukturen für oberflächenverstärkte Raman-Spektroskopie.
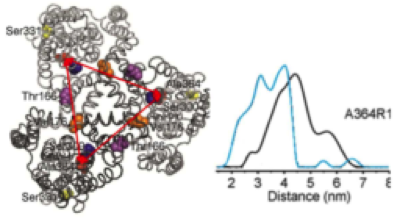
Mittels DEER ermittelte Abstandsverteilung des Aspartattransporters Glt(Ph) in An- (blau) und Abwesenheit (schwarz) von Substrat (Hänelt et al., Nat. Struct. Mol. Biol., 2013)
AG Heinz-Jürgen Steinhoff, Physik
Makromolekülstruktur - ESR-Spektroskopie von Membranproteinen
In der AG Steinhoff kooperieren Physiker/Innen, Chemiker/Innen und Biolog/Innen interdisziplinär. Wir charakterisieren die Struktur und Dynamik biologisch oder medizinisch relevanter Makromoleküle und ihrer Komplexe. Ziel ist das Verständnis ihrer Funktion auf atomarer Ebene. Dazu werden die Elektronenspinresonanz-Spektroskopie und Spinmarkierungstechniken methodisch weiterentwickelt und mit Molekulardynamik-Simulationen komplementiert. Im Fokus stehen die Funktionsmechanismen des transmembranen Substrattransports und Signaltransfers.
AG Christian Ungermann, Biologie
Membrandynamik im endosomalen Weg
Die AG Ungermann ist an der Fusion von Membranen im endolysosomalen Weg der Hefe und höherer Eukaryonten interessiert. Die Gruppe hat Protokolle zur Reinigung und biochemischen Charakterisierung bis hin zur Strukturanalyse entwickelt, um die Funktion von großen Proteinkomplexen an Endosomen und Vakuolen analysieren zu können, die an Fusion und Membranremodellierung beteiligt sind. Sie etabliert neue in vitro Assays zur Vermessung der Anbindung von Membranen und anschließenden Fusion und hat Erfahrungen mit Liposomen-basierten Interaktionsanalysen. Zusätzlich fokussiert sich die Gruppe auf die in vivo Analyse von Endosomen und Vakuolen und deren Assoziationen mit Proteinkomplexen, um deren Dynamik und Funktion zu klären.
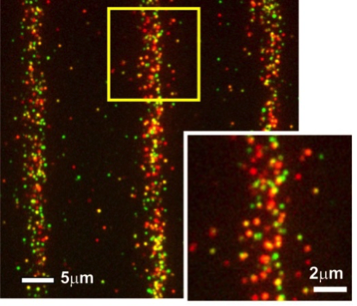
Einzelzell pull-down Test von individuellen, zytosolischen STAT1 (rot)/STAT2 (grün) Komplexen auf einer mikrostrukturiert funktionalisierten Oberfläche (Wedeking et al., Nano Lett. 2015).
AG Changjiang You, Biologie
Biofunktionalisierung von Oberflächen zur Quantifizierung und Manipulation von zellulärer Signalvermittlung
Die Nachwuchsgruppe You biofunktionalisiert Nanopartikel und planare Substrate zur funktionalen Immobilisierung und lateralen Organisation von Proteinen an Oberflächen. Eine neuartige Monofunktionalisierung von quantum dots wurde erfolgreich implementiert. Sie erlaubt die Untersuchung molekularer und zellulärer Determinanten der Signalaktivierung über Zytokinrezeptoren auf Einzelmolekülniveau. Mittels mkrostrukturiert biofunktionalisierter Substrate können wir die Stöchiometrie und Dynamik von Proteinkomplexen in der Signaltransduktion von Zytokinrezeptoren quantifizieren. Derzeit werden diese Methoden weiterentwickelt, um die raum/zeitliche Organisation von Signalkomplexen in der Plasmamembran lebender Zellen kontrollieren zu können.

