Hauptinhalt
Topinformationen
Bildanalyse, Modellierung und Simulation
Koordinator: Stefan Kunis
Forschungsfeld D zielt auf die Entwicklung von Algorithmen und Methoden anhand konkreter biologischer Fragestellungen in enger Zusammenarbeit mit Arbeitsgruppen aus der Mathematik, Physik und den Kognitionswissenschaften. Insbesondere sollen
- Daten aus bildgebenden Verfahren analysiert, dargestellt und quantitativ ausgewertet werden
- kausale Zusammenhänge und Korrelationen in komplexen Daten identifiziert werden
- molekulare Prozesse bis hin zu vollständigen Mikrokompartimenten mechanistisch beschrieben werden.
Beteiligte Wissenschaftlerinnen und -wissenschaftler
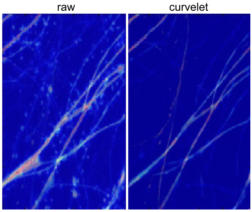
Morphologie von Neuronen in Hirnschnitten transgener Mäuse. Links: Originalbild; rechts: Dendritischer Rückgrat nach Curvelet-Analyse (AG Brandt, AG Kutyniok, unveröffentlicht).
AG Roland Brandt, Biologie
Mechanismen der Entwicklung und Degeneration von Nervenzellen
Thema der AG Brandt ist die Aufklärung der molekularen Mechanismen der Entwicklung, des Alterns und der Degeneration von Nervenzellen. Einer der Schwerpunkte der Arbeitsgruppe besteht in der Entwicklung und dem Einsatz quantitativer „live cell imaging“ Methoden, um die Dynamik von Komponenten des neuronalen Zytoskeletts zu verfolgen. Die Analyse erfolgt in zellulären Modellen und organotypischen Hirnkulturen. Darüber hinaus entwickelt die Arbeitsgruppe neue Mausmodelle, um Mithilfe von konfokaler und 2-Photonen-Mikroskopie morphologische Veränderungen von Nervenzellen in einem systemischen Kontext zu bestimmen.
AG Mirco Imlau, Physik
Ultraschnelle, nichtlinear-optische Spektroskopie
Die AG Imlau beschäftigt sich mit der ultraschnellen, nichtlinear optischen Spektroskopie an photofunktionalen Dielektrika vor dem Hintergrund der atomaren Kontrolle von Licht mittels Licht. Die Gruppe entwickelt hierzu spektroskopische Methoden auf Basis der Holographie, um lichtinduzierte Änderungen der komplexen Permittivität mit hoher Zeitauflösung über einen großen Frequenzbereich zu erfassen. In diesem Zusammenhang werden die ultraschnellen Eigenschaften von kleinen gebundenen Polaronen in Festkörperkristallen bzw. von Bindungsisomerien in Übergangsmetallkomplexverbindungen untersucht und weiterführende Konzepte für die nichtlineare Wechselwirkung von Licht mit Materie entwickelt.

Vergleich der Blickbewegungen (rechts) mit Modellvorhersagen (Mitte) für natürliche Stimuli (links) (Wilming et al., PLoS One 2011)
AG Peter König, Kognitionswissenschaft
Neurobiologische Grundlagen kognitiver Funktionen - Visuelle Verarbeitung und Embodiment
Die Arbeitsgruppe König untersucht die multisensorische Integration und sensorimotorische Kopplung. Dafür setzt sie sowohl experimentelle als auch theoretische Methoden ein. Diese beinhalteten zum einen die quantitative Auswertung des subjektiven Erlebens, Messung von Augenbewegungen, EEG, MEG und fMRI. Zum anderen erfolgt die Modellierung neuronaler Systeme im Kontext von aktiver Wahrnehmung und der Verarbeitung natürlicher Reize mit neuronalen Netzwerken und hierarchischen Bayes-Modellen. In engem Wechselspiel experimenteller und theoretischer Methoden werden Hypothesen überprüft und verfeinert. Die Ergebnisse unserer Arbeiten werden, so weit möglich, in Anwendungen übertragen. Dies hat zur Gründung von zwei Spin-off Firmen geführt.
AG Stefan Kunis, Mathematik
Analysis, Algorithmen, Signal- und Bildverarbeitung
Die AG Kunis arbeitet in der angewandten und numerischen Analysis. Insbesondere beschäftigt sich die Gruppe dabei mit Methoden der harmonischen Analysis und Approximationstheorie zur Konstruktion schneller und robuster Algorithmen für hochdimensionale Probleme und deren effiziente Implementierung. Den Schwerpunkt bilden hierbei verallgemeinerte Fourier-Transformationen. Die bisher bearbeiteten Anwendungen sind neuartige Algorithmen für tomographische Bildgebungsverfahren, in jüngster Vergangenheit insbesondere für die photoakustische Tomographie.
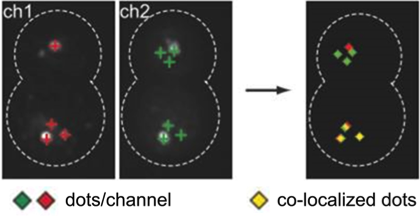
Automatisierte Einzelzellkolokalisationsanalyse (ImageJ plugin) am Beispiel von Endosommarkern in Hefezellen (Arlt et al., Mol Biol Cell 2015)
AG Rainer Kurre, Biologie
Prozessierung und Visualisierung komplexer mehrdimensionaler Mikroskopiedaten
Komplexe mehrdimensionale Datensätze aus der Mikroskopie müssen generell aufbereitet und weiterverarbeitet werden, bevor sie in geeigneter Weise visualisiert werden können. Wir bieten verschiedene Techniken der Dekonvolution sowie Volumen und Oberflächen-Renderung an, um dynamische Prozesse mit höchstem Kontrast darzustellen. Desweiteren werden für verschiedenste biologische Fragestellungen (Protein-Protein-Kolokalisation, Quantifizierung morphologischer Veränderungen, digitale Segmentierung, usw.) maßgeschneiderte Analysewerkzeuge entwickelt und in etablierten Softwarelösungen wie z.B. ImageJ oder Matlab integriert.
AG Phillip Maaß, Physik
Statistische Physik
Die AG Maaß beschäftigt sich mit Strukturbildung fern vom Gleichgewicht, mit Transport- und Relaxationsprozessen in kondensierter Materie und mit grundlegenden Fragen der Statistischen Physik des Nichtgleichgewichts. Hierbei werden analytische Verfahren, numerische Methoden und Computersimulationen in Kombination eingesetzt. Untersuchungen zur Wachstumskinetik auf Oberflächen, zum Ionentransport in Festelektrolyten, zur Thermodynamik nanoskopischer Maschinen, zur Struktur und Dynamik von Lipidmolekül-Wasser-Komplexen, zur Dichtefunktionaltheorie von Gitterfluiden und zum getriebenen Teilchentransport in Kanalstrukturen bilden Schwerpunkte der derzeitigen Forschungstätigkeit.
AG Armen Mulkidjanian, Physik
Bioinformatik
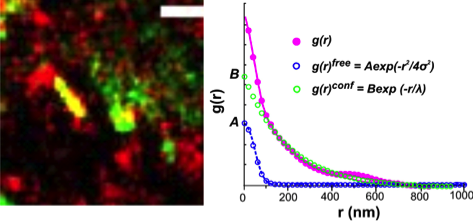
Paar-Korrelations-Analyse von Einzelmolekülverfolgung und -Lokalisationsmikroskopie (pcTALM) der Rekrutierung eines Effektors (rot) an Quantum Dot-markierte Zytokinrezeptorkomplexe (grün, You et al., Anal Chem 2014)
AG Jacob Piehler, Biologie
Signalvermittlung über biologische Membranen - Bildverarbeitung für die Einzelmolekülmikroskopie
Für die Analyse von Bilddaten aus der Einzelmolekül-Lokalisationsmikroskopie hat die AG Piehler eine integrierte Auswertungssoftware entwickelt. Neben der Erstellung und Analyse höchstaufgelöster Bilder stehen insbesondere Algorithmen zur Quantifizierung von Diffusionsdynamik und von Wechselwirkungen mittels räumlicher und zeitlicher Korrelation einzelner Moleküle im Fokus. In Zusammenarbeit mit der Mathematik weiten wir diese Arbeiten darauf aus, über Einzelmoleküllokalisation bzw. strukturierte Beleuchtung schnelle höchstaufgelöste 3D Bildgebung zu erreichen.
AG Gordon Pipa, Kognitionswissenschaften
Statistische Modell- /Hypothesenbildung
Die AG Pipa beschäftigt sich mit der Modellierung experimentell beobachteter dynamischer Prozesse, um kausale Zusammenhänge in Messdaten zu erkennen und diese durch Netzwerke zu abstrahieren und zu visualisieren. Ziel dieser Modellierung ist es Strukturen in komplexen Interaktionen zu erkennen und damit Forschungshypothesen zu generieren, zu unterstützen oder zu wiederlegen. Um solche Zusammenhänge aus komplexen Datenstrukturen zu extrahieren, nutzt die AG Pipa effiziente Methoden aus dem Bereich des maschinellen Lernens, die sich bereits auf sehr komplexen Datensätzen aus den Kognitionswissenschaften bewährt haben.

